BELÉN ELGUERO 1, DAVID GONILSKI PACIN 1, CAROLINA CÁRDENAS FIGUEROA 1, MARIANA FUERTES 1, EDUARDO ARZT 1, 2
1 Instituto de Investigación en Biomedicina de Buenos Aires (IBioBA) – CONICET – Partner Institute of the Max Planck Society, 2 Departamento de Fisiología y Biología Molecular y Celular, Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires, Argentina
Resumen Las modificaciones postraduccionales (PTMs por su nombre en inglés) son modificaciones covalentes en las proteínas durante o posteriores a su síntesis. Las más conocidas son fosforilación, metilación y acetilación, también clivajes o unión de pequeños péptidos (ubiquitinación, SUMOilación y NEDDilación). Frecuentemente la proteína es modificada en múltiples sitios y estas modificaciones se coordinan generando una interacción de PTMs. Patrones alterados de PTMs han sido relacionados con varias enfermedades. En la actualidad los avances en la espectrometría de masas han hecho posible estudiar en simultáneo múltiples PTMs. La oncología es una de las disciplinas que ha incorporado estas tecnologías por su necesidad de caracterizar a los tumores. En cáncer se han descripto varias alteraciones relacionadas a las PTMs del tipo ubiquitina, como la SUMOilación. En particular la interacción entre distintas PTMs con la SUMOilación ha sido estudiada en el contexto de la enfermedad multitumoral de von Hippel Lindau, generando posibles nuevos biomarcadores para la evolución de estos tumores. RSUME o RWDD3, un enhancer de SUMOilación que actúa sobre las proteínas VHL y HIF, ha mostrado una correlación con parámetros malignos en este tipo de tumores, como la angiogénesis. Los reguladores de las PTMs están cobrando relevancia como biomarcadores en el cáncer.
Palabras clave: modificaciones postraduccionales, cáncer, biomarcador, SUMOilación, RSUME/RWDD3
Abstract Post-translational modifications (PTMs) are covalent modifications in proteins during or after their synthesis. Among them, the best known are phosphorylation, methylation, acetylation, and also cleavage or binding of small peptides (ubiquitination, SUMOylation and NEDDylation). Often the protein is modified in multiple sites and these modifications are coordinated generating a PTMs crosstalk. Altered patterns of PTMs have been related to several pathologies. Currently, advances in mass spectrometry have made it possible to study multiple PTMs simultaneously. Oncology is one of the disciplines that incorporated these technologies for the need to better characterize tumors. In cancer, several alterations related to the ubiquitin-like PTMs have been described, such as SUMOylation. In particular, the interaction between different PTMs with SUMOylation has been studied in the context of the von Hippel Lindau (VHL) multitumoral syndrome, generating new putative biomarkers for the evolution of these tumors. RSUME or RWDD3, an enhancer of SUMOylation that acts on VHL and HIF proteins, shows a correlation with malignant parameters in this type of tumors, such as angiogenesis. Regulators of PTMs are becoming relevant as biomarkers in cancer.
Key words: post-translational modifications, cancer, biomarker, SUMOylation, RSUME/RWDD3
Dirección postal: Eduardo Arzt, Godoy Cruz 2390, 1425 Buenos Aires, Argentina
e-mail: earzt@ibioba-mpsp-conicet.gov.ar
Las modificaciones postraduccionales (PTMs por su nombre en inglés) son modificaciones covalentes que ocurren a lo largo de la vida de una proteína, en las cadenas laterales de sus aminoácidos o en los extremos de las mismas. Estos cambios pueden tomar distintas formas: pequeñas modificaciones químicas como son fosforilación, metilación y acetilación; también incluyen clivajes o unión de pequeños péptidos (ubiquitinación, SUMOilación y NEDDilación) 1. Es por ello que pueden ser consideradas como la última etapa de la expresión génica y, más importante aún, permiten diversificar el repertorio de proteínas posibles de generar. A su vez nos permiten comprender la gran discrepancia entre el genotipo y la gran diversidad de fenotipos observados, tanto en condiciones fisiológicas como en enfermedades.
Las PTMs pueden clasificarse en irreversibles (clivajes proteolíticos, oxidación de cisteína, etc.) o reversibles (fosforilación, metilación, ubiquitinación y SUMOilación, entre otras). Estas últimas son particularmente interesantes ya que se regulan dinámicamente frente a condiciones específicas y al entorno celular, permitiendo modificar la función, las interacciones con ADN, otras proteínas y macromoléculas, así como también la actividad, localización subcelular y su ensamblaje en complejos de mayor orden de las proteínas sobre las que ocurren 1, 2. Es importante aclarar que históricamente se consideraban PTMs reversibles a aquellas catalizadas por enzimas e irreversibles a las que ocurren como respuesta de reacciones químicas espontaneas, como puede ser la oxidación de cisteína a ácido sulfónico. Sin embargo, se sabe que muchas de estas reacciones no enzimáticas, anteriormente consideradas irreversibles, son reguladas por el estado redox celular (para citar un ejemplo) y son, por lo tanto, reversibles, reflejando un estado particular de la célula y activando respuestas frente al mismo.
Hasta la fecha se han encontrado más de 200 PTMs en humanos 3. Si bien el estudio de las mismas ocurre en una primera etapa a nivel de la modificación individual en un residuo de la proteína, frecuentemente la proteína es modificada en múltiples sitios, generando un código en el que, a través de interacciones positivas y negativas, estas modificaciones se coordinan para determinar la respuesta resultante, proceso llamado interacción de PTMs, como ocurre en el claro ejemplo del código de histonas (PTM crosstalk) 2.
Modificaciones postraduccionales en la medicina y su relevancia en diferentes enfermedades
Como consecuencia de la variabilidad entre individuos en respuesta a la terapia, la clínica médica ha cambiado su enfoque hacia la medicina personalizada. Tanto esta última como la investigación aplicada se centran en encontrar la conexión/relación entre el genotipo y el fenotipo. Desde esta perspectiva surge el estudio del proteoma, conjunto de proteínas que una célula posee al momento del análisis, que es en última instancia el que define el fenotipo.
Para una proteína dada la combinación de PTMs genera una variante diferente de la original y define la función, el proteoma incluye como variantes todas aquellas proteínas que poseen diferentes PTMs. Patrones alterados de PTMs han sido relacionados con varias afecciones, ya sean la causa o el resultado, lo que los vuelve blancos relevantes para estudios de biomarcadores y de patogénesis de las mismas 1, 4. En la actualidad, los avances en la espectrometría de masas (MS por sus siglas en inglés), técnica que permite separar e identificar proteínas o fragmentos de ellas, ha hecho posible estudiar en simultáneo múltiples PTMs a partir de pequeñas cantidades de muestra. Por ello es que este nuevo enfoque, la PTMomica cuantitativa, tiene un gran potencial para descubrir biomarcadores a partir de fluidos corporales y de tejidos, así como también dilucidar los mecanismos de la enfermedad a través de la caracterización de las vías de señalización 1. Este estudio también abarca herramientas bioinformáticas para la identificación de nuevos PTMs y la interpretación de resultados.
Se han desarrollado numerosas bases de datos (revisadas en Pascovici, 2018) que pueden clasificarse en categorías como aquellas utilizadas para la localización y predicción de sitios posibles de sufrir modificaciones, para predicción de la función e interacción de una proteína modificada y para visualización de la estructura proteica resultante de una PTM 5.
Entre las modificaciones postraduccionales relacionadas con enfermedades y estudiadas en muestras clínicas se encuentran la fosforilación asociada a Alzheimer, diabetes y cáncer; la glicosilación que juega un rol importante en cáncer, en enfermedades neurodegenerativas e inflamación; el clivaje proteolítico que se ha vinculado a enfermedades cardiovasculares, neurodegenerativas, inflamación y cáncer 4; y la ubiquitinación también asociada enfermedades autoinmunes, neurodegenerativas y cáncer 5.
Modificaciones postraduccionales y cáncer
La relevancia de estudiar PTMs en cáncer, adicionalmente de la búsqueda de biomarcadores para detección temprana y de la progresión del mismo 5, radica en poder seleccionar la estrategia terapéutica apropiada para el tratamiento de las alteraciones moleculares subyacentes a un tipo de tumor. Dichas alteraciones dependerán de la clasificación inicial y de la historia natural del tumor, que involucra la heterogeneidad de poblaciones celulares, fisiología del paciente y los diferentes tratamientos a los que fue expuesto. En algunos casos estas modificaciones están determinadas por mutaciones, situación en que el enfoque genómico brindará información importante pero, a su vez, el espectro de mutaciones complejas y heterogéneas hace difícil predecir si la presencia de anomalías genéticas se traducirá en cambios de expresión génica y en el fenotipo de células. Además, el tumor responde dinámicamente a un entorno constantemente cambiante como es el microambiente tumoral, a través de la regulación de la expresión génica dada, en última instancia, por PTMs. Es por ello que la investigación en oncología rápidamente ha incorporado los recientes avances en tecnologías asociadas a proteómica 5. Un ejemplo notable es la aprobación por la FDA (Food and Drug Administration) de PTMs como biomarcadores en plasma de pacientes con glioblastoma 6.
Ubiquitina y Ubls (proteínas de la familia de las ubiquitinas) en cáncer
La ubiquitinación y conjugación de péptidos de tipo ubiquitina (Ubl) involucran una clase de péptidos con servados evolutivamente que se conjugan en forma covalente a proteínas de forma reversible y que afectan a casi todos los aspectos de la regulación celular 3. Estos incluyen ubiquitina (Ub), pequeño modificador similar a la ubiquitina (SUMO por su nombre en inglés), precursor expresado por células neurales, regulado negativamente durante el desarrollo 8 (NEDD8 por su nombre en inglés), proteína 8 relacionada con la autofagia (ATG8 por su nombre en inglés), proteína 12 relacionada con la autofagia (ATG12 por su nombre en inglés), HLA-F adjacent transcript 10 (FAT10), y gen 15 estimulado por interferón (ISG15 por su nombre en inglés), entre otros 7. Las vías de conjugación de estas pequeñas etiquetas peptídicas requieren una cascada de reacciones llevada a cabo por enzimas activadoras de Ub o Ubls (E1), de conjugación (E2) y ligasas (E3). Estos tres tipos de enzimas actúan secuencialmente. La etiqueta peptídica se activa primero por medio de una E1 y luego se transfiere a una enzima conjugadora E2. Bajo ciertas circunstancias, E2 puede dirigir la conjugación a la proteína diana, pero la mayoría de las veces requiere la actividad cooperativa de las ligasas E3, que interactúan al mismo tiempo con una E2 y con la proteína sustrato 3. Las distintas modificaciones con Ubls tienen roles diferentes. En el proceso de ubiquitinación, dependiendo del enlace Ub, las cadenas poliUb pueden marcar a las proteínas para su degradación a través del proteasoma 26S, o pueden actuar como intermediarios para la formación de complejos de proteínas. En contraste, la conjugación con una sola Ub (monoubiquitinación) puede regular la actividad o localización de las proteínas.
A su vez la conjugación con el péptido SUMO se asocia con la regulación de la actividad proteica, la formación de complejos, la localización nuclear o citoplásmica y la transcripción. Mientras que el péptido NEDD8 se conjuga principalmente con proteínas cullinas para activarlas para la conjugación Ub, las Ubls ISG15 y FAT10 juegan un papel en la respuesta inmune innata. Otros Ubls notables son ATG8 y ATG12, que funcionan en la autofagia 7 (Fig. 1).
Como consecuencia de la diversidad de funciones de este tipo de PTMs, las alteraciones en las vías asociadas a Ub y Ubl se han implicado en la patogénesis de numerosas enfermedades humanas, como el cáncer. Se ha reportado sobreexpresión de varias E3 ligasas de Ub en la leucemia mieloide aguda (WWP1), carcinoma de células renales (UBE3C), linfoma y cáncer de mama, páncreas, colon y próstata (Ubc13)5. También se ha relacionado la expresión de enzimas involucradas en la NEDDilación (por ejemplo: NEDD8 E1, NAE1/UBA3 y NEDD8 E2, BE2M/UBE2F) con cáncer de pulmón, colorrectal e hígado, colangiocarcinoma intra-hepático, glioblastoma y carcinoma nasofaríngeo y de células escamosas esofágicas 8.
Actualmente hay numerosos agentes quimioterápicos enfocados en estas PTMs que están siendo desarrollados o que ya han sido aprobados y están en pruebas en fase clínica. Si bien inhibidores del proteasoma (bortezomib/velcade/PS-341 y carfizomib) son utilizados en la clínica, son inespecíficos y pueden provocar efectos secundarios.
En contraste, la especificidad de sustrato de las enzimas conjugadoras de Ubls las hace un objetivo atractivo para el tratamiento3. En el caso de inhibidores de actividad E1, el único que está en pruebas de fase II es el MLN4924 (inhibidor de E1 de NEDD8) ya que las E1 de Ub también muestran inespecificidad parecida a los inhibidores del proteasoma9. En oposición, debido a la mayor especificidad de las ligasas E3, se han desarrollado múltiples drogas basadas en estas enzimas. GDC-199 (venetoclax) es un compuesto que inhibe la actividad SCFSKP2, una E3 ligasa de reguladores del ciclo celular como p27Kip1 y p21/Cip1, que posee buenos resultados en pruebas con animales 9. Nutlins es un análogo de cis-imidazolina que inhibe MDM2 (E3 ligasa de p53), mientras que MI-219 inhibe la interacción entre MDM2 y p53, ambos están siendo probados actualmente en fase clínica 9. En la actualidad, el desarrollo de quimioterápicos relacionados con el sistema Ub está orientado a compuestos conocidos como PROTACs (moléculas quiméricas dirigidas a proteínas), basado en el concepto de generar moléculas artificiales para reclutar una Ub ligasa específica a una proteína objetivo, no haciendo blanco en la desregulación de esta vía de ubiquitinación sino más bien utilizándola como herramienta.
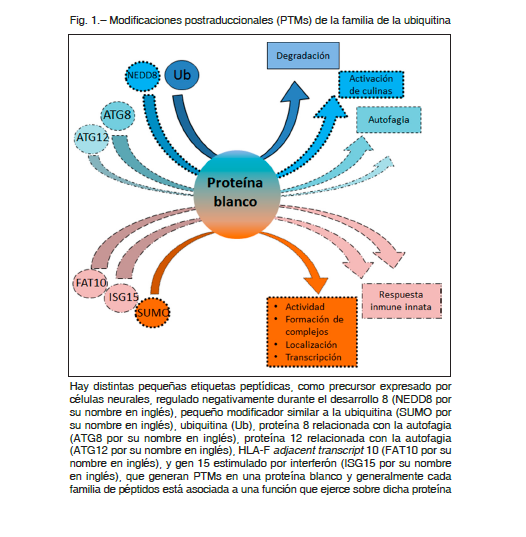
Rol de la SUMOilación en el cáncer
La SUMOilación modula una diversidad de procesos biológicos cuya desregulación está relacionada con la patogénesis del cáncer, como la reparación del daño en el ADN, progresión del ciclo celular y apoptosis, así como también procesos que afectan la progresión tumoral como la repuesta inmune, la regulación de la división mitocondrial, la regulación de canales de voltaje y ritmos biológicos.
Los componentes de la vía de SUMOilación incluyen los cuatro parálogos del péptido SUMO (SUMO1, SUMO2, SUMO3 y SUMO4), los cuales requieren de un clivaje por proteínas proteasas específicas de setrina (SENPs, por su nombre en inglés) para ser activos. También incluye a enzimas activadoras (SAE1 y SAE2) que transfieren el péptido a la enzima siguiente llamada conjugadora Ubc9, y esta última une el péptido al sustrato (hasta la fecha solo se ha descripto solo una enzima conjugadora, Ubc9).
A su vez hay enzimas E3 ligasas (PIAS, RanBP2 y Pc2, entre otras) que favorecen la conjugación y su expresión se ha hallado elevada en varias neoplasias malignas y se asocia con un mal pronóstico de la enfermedad. Varios grupos han proporcionado evidencia de que la inhibición de la vía SUMO a través de la disminución en la expresión de SAE o Ubc9 inhibe el crecimiento de tumores en modelos de ratón 10. Además, la reducción de SAE confiere letalidad sintética (reducción en la viabilidad de la célula tumoral por adicionar una segunda mutación o cambio en la expresión de un gen cuando hay mutaciones genéticas determinadas en las células malignas) en tumores con alta actividad de MYC o mutaciones KRAS, si bien se han reportado varios inhibidores de Ubc9 (GSK145A, 2-D08 y spectomicina B1) su especificidad es poco clara y parecen tener baja potencia 10. Sin embargo, dada la relevancia de esta PTM en cáncer, la búsqueda de nuevos inhibidores de estas vías sigue siendo un campo muy activo. Comparada a la ubiquitinación, la SUMOilación en general confiere mayor estabilidad, y su principal característica es modificar la superficie de sus proteínas blanco para regular las interacciones proteína-proteína, y así mediar la localización y función de las mismas. Considerando esto, una alternativa al enfoque de estudiar los componentes involucrados en el procesamiento de esta PTM es el estudio de moléculas que interactúan con las diferentes enzimas de esta vía.
Rol de RSUME en el cáncer
RSUME (RWD-containing sumoylation enhancer) o RWDD3 es una proteína identificada a partir de una línea celular hipofisaria tumoral con capacidad angiogénica aumentada, que actúa como estimulador de la cascada de SUMOilación a través de una interacción directa con la enzima conjugadora de SUMO Ubc9 11. Esta proteína posee un dominio RWD (RING finger and WD repeat containing proteins and DEAD-like helicases) que es esencial para su actividad y, si bien ejerce su efecto durante la formación del enlace tio-éster Ubc9-SUMO-1, también actúa en la transferencia de SUMO-1 del enlace tio-éster a un sustrato específico. Se la encontró expresada gliomas y tumores de hipófisis y páncreas, entre otros 12,13. Además, también se ha identificado en tumores de mama con alto riesgo de recurrencia y metástasis 14. RSUME está íntimamente conectado con el síndrome multitumoral hereditario de von Hippel Lindau, causado por mutaciones en el gen de VHL, ya que se expresa en órganos con predisposición a la formación de tumores VHL y en hemangioblastomas y feocromocitomas, característicos de esta enfermedad 15.
Un análisis bioinformático realizado por The Human Protein Atlas de un reciente conjunto de datos del cáncer Genome Atlas Research Network, muestra que el 20.1% de 528 muestras estudiadas de tumores de cáncer renal de células claras, una de las principales causas de muerte de pacientes VHL, expresa niveles elevados de RSUME, y se correlaciona con una disminución del 23% en la supervivencia de los pacientes 16
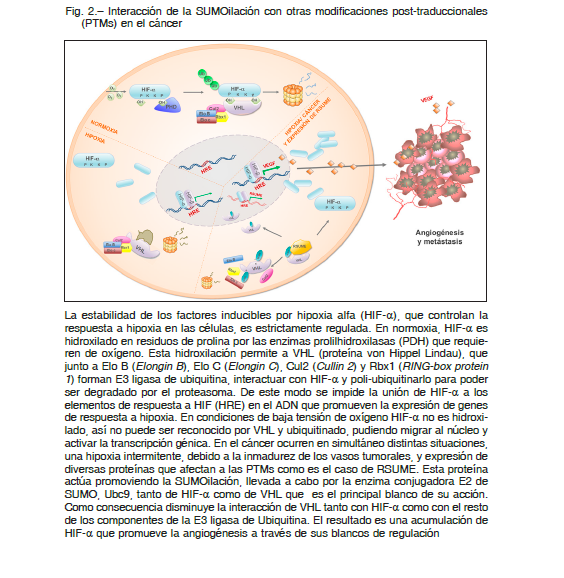
La proteína VHL es un supresor tumoral, pero resulta interesante que forme parte de un complejo E3 ligasa de Ub, cuyo blanco más estudiado en cáncer es el factor inducible por hipoxia alfa (HIF-α), proteína que juega un rol muy importante en promover la angiogénesis tumoral.
Recientemente, se ha reportado a RSUME como promotor de este proceso, lo que lo vuelve un blanco interesante de futuras terapias, como así también un biomarcador en estos tipos de cáncer 16 (Fig. 2).
En condiciones normales de oxígeno, enzimas llamadas prolil hidroxilasas (PDH), catalizan la hidroxilación de prolinas, luego este residuo así modificado es reconocido y puede interactuar con VHL, que opera como el elemento de reconocimiento de un complejo Ub ligasa E3, que conduce a la destrucción final de HIF-α por el proteasoma.
A su vez, la SUMOilación afecta a varios componentes de este sistema, la hipoxia a través del aumento de la expresión de RSUME (que actúa como enhancer de este proceso) incrementa la SUMOilación de HIF y VHL. En el caso de HIF-α la sumoilación aumenta su estabilidad, mientras que la SUMOilación de VHL regula su distribución espacial y disminuye su interacción con HIF-α 16.
Este sistema de regulación que involucra a VHL, HIF-α y RSUME, es un ejemplo de múltiples nodos de regulación que ocurren en procesos celulares e ilustra claramente la interrelación entre las diversas PTMs frente a la respuesta celular inducida por estímulos internos y externos. El mayor conocimiento de estas interconexiones es una herramienta importante para definir nuevos biomarcadores para el cáncer basados en proteómica y PTMomica, como así también para el desarrollo de nuevas estrategias terapéuticas.
Agradecimientos: Este trabajo fue financiado por la Universidad de Buenos Aires (UBA), el Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET) y la Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT) de Argentina, y el Fondo para la Convergencia Estructural del Mercosur (FOCEM) (COF 03/11).
Conflicto de intereses: Ninguno para declarar
Bibliografía
1. Thygesen C, Boll I, Finsen B, Modzel M, Larsen MR. Characterizing disease-associated changes in post-translational modifications by mass spectrometry. Expert Rev Proteomics 2018; 15: 245-58.
2. Csizmok V, Forman-Kay JD. Complex regulatory mechanisms mediated by the interplay of multiple post-translational modifications. Curr Opin Struct Biol 2018; 48: 58-67.
3. Eisenberg-Lerner A, Ciechanover A, Merbl Y. Post-translational modification profiling – A novel tool for mapping the protein modification landscape in cancer. Exp Biol Med (Maywood) 2016; 241: 1475-82.
4. Pagel O, Loroch S, Sickmann A, Zahedi RP. Current strategies and findings in clinically relevant post-translational modification-specific proteomics. Expert Rev Proteomics 2015; 12: 235-53.
5. Pascovici D, Wu JX, McKay MJ, et al. Clinically relevant posttranslational modification analyses-maturing workflows and bioinformatics tools. Int J Mol Sci 2018; 20: pii: E16.
6. Petushkova NA, Zgoda VG, Pyatnitskiy MA, et al. Posttranslational modifications of FDA-approved plasma biomarkers in glioblastoma samples. PLoS One 2017; 12: e0177427.
7. Streich FC, Jr., Lima CD. Structural and functional insights to ubiquitin-like protein conjugation. Annu Rev Biophys 2014; 43: 357-79.
8. Zhou L, Zhang W, Sun Y, Jia L. Protein neddylation and its alterations in human cancers for targeted therapy. Cell Signal 2018; 44: 92-102.
9. Huang, X, Dixit VM. Drugging the undruggables: exploring the ubiquitin system for drug development. Cell Res 2016; 26: 484-98.
10. He X, Riceberg J, Soucy T, et al. Probing the roles of SUMOylation in cancer cell biology by using a selective SAE inhibitor. Nat Chem Bio 2017; 13: 1164-71.
11. Carbia-Nagashima A, Gerez J, Perez-Castro C, et al. RSUME, a small RWD-containing protein, enhances
SUMO conjugation and stabilizes HIF-1alpha during hypoxia. Cell 2007; 131: 309-23.
12. Chen X, Kuang W, Huang H, et al. Knockdown of RWD domain containing 3 inhibits the malignant phenotypes of glioblastoma cells via inhibition of phosphoinositide 3-kinase/protein kinase B signaling. Exp Ther Med 2018; 16: 384-93.
13. Wu Y, Tedesco L, Lucia K, et al. RSUME is implicated in tumorigenesis and metastasis of pancreatic neuroendocrine tumors. Oncotarget 2016; 7: 57878-93.
14. Huang CC, Tu SH, Lien HH, et al. Concurrent gene signatures for han chinese breast cancers. PLoS One 2013; 8: e76421.
15. Gerez J, Tedesco L, Bonfiglio JJ, et al. RSUME inhibits VHL and regulates its tumor suppressor function. Oncogene 2015; 34: 4855-66.
16. Tedesco L, Elguero B, Pacin DG, et al. von Hippel-Lindau mutants in renal cell carcinoma are regulated by increased expression of RSUME. Cell Death Dis 2019; 10: 266.
